M20 Genomics在全球范围内首发单细菌转录组测序平台MscRNA-seq,开启单细菌测序新篇章
M20 Genomics在全球范围内首次正式发布单细菌转录组测序的商业平台MscRNA-seq (全称:Microbial-single-cell RNA sequencing)。这意味着在微生物检测领域中,单细菌转录组测序技术迈入商业化阶段,将对研究细菌的耐药、致病、持留及细菌与宿主的相互作用等方面的研究起到巨大的推动作用。
单细菌测序技术的现状
单细胞测序技术可以在单个细胞的水平上对细胞的基因表达等信息进行检测,解决了传统研究以组织或细胞群作为样本进行测序时,单个细胞与细胞之间的差异有可能被群体的平均值所掩盖的问题。
长期以来,细菌耐药性问题严重。根据世卫组织(WHO)2014年发表的全球抗生素耐药报告显示,当年全球至少有70万人死于耐药细菌感染,并且预测到2050年全球每年将有1000万人以上死于耐药细菌的感染,超过当年因癌症死亡人数820万。近年来,在新的抗生素技术没有重大突破的现状下,每年死于耐药细菌感染的人数在不断上升,保守估计已经突破百万。
单个细菌之间的耐药性存在巨大的个体差异性,通过传统高通量测序方法对细菌耐药性展开研究时,很难解决这一问题。随着单细胞测序技术的发展,研究者们希望能将这一技术应用到细菌上,以展开更加深入的研究。
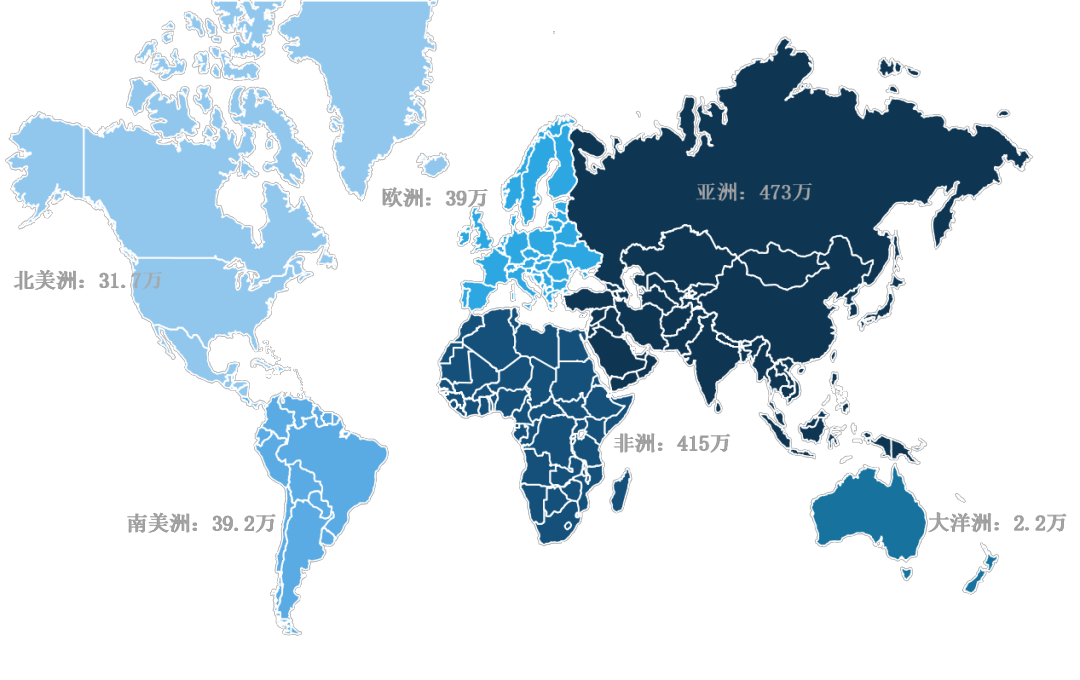
(2050年全球因感染超级耐药细菌死亡人数预测,数据来自《抗菌素耐药:全球监测报告》, WHO, 2014)
目前,一些应用单细菌基因组技术的研究已经展开,有研究表明,单细菌基因组测序对未培养的微生物有着极高的分辨率,通过这项技术研究人员可一次性获得5万个以上细菌的数据,从而了解微生物群落中抗生素抗性基因、毒性因子等特征的分布情况[1]。
此外,单细菌基因组技术还可应用于微生物的分类和功能注释,新分支和物种的发现,帮助研究者探索微生物亚种之间的差异。

(显微镜下的大肠杆菌)
单细菌RNA测序将开启细菌研究的新阶段
虽然单细菌基因组测序已在一些研究中有所应用,但是在单细菌转录组测序技术上,受到细胞尺寸小,细胞分离困难、细胞壁的存在、mRNA分子缺乏polyA尾等因素的影响,现有的技术难以直接应用到细菌等微生物上。
10X Genomics作为目前全球最大的单细胞测序商业平台之一,自2016年以来发布了一系列单细胞测序相关产品,但其平台并不支持单细菌转录组测序。
为满足单细菌RNA测序的需求,有科研人员发表了针对原核细胞的单细胞RNA测序方法PETRI-seq和microSPLiT,分别刊登在Nature Microbiology[2]和Science[3]上。两种技术手段原理类似,均是通过Split-pool的方式实现单个细菌细胞的标记,来完成原核细胞的单细胞RNA测序。
但是Split-pool方法的稳定性较低,取得的基因数也较少,目前很难进行商业自动化的应用。因此,开发适合商业自动化的单细菌转录组测序技术将非常有必要。
M20 Genomics的单细菌测序平台:成为领航者
(M20开发的新一代 单细胞测序仪)
在M20研发的新一代单细胞测序文库构建平台上,我们实现了细菌的高通量单细胞RNA测序,并获得了非常好的检测结果。目前已经检测过大肠杆菌、鲍曼不动杆菌、肺炎克雷伯菌等多种类型的细菌,大部分样本的单个细菌中可以检测到上千个基因,助力了细菌新的耐药和持留机制的研究发现。
通过单细菌测序技术,可以对无法在实验室培养的菌群进行深度测序,或者直接进行感染的早期诊断和耐药检测,并能够挖掘出一些环境微生物,其基因和通路能够为生物技术和医学所用。
M20单细菌转录组测序平台的发布打破了单细胞测序的核心关键技术长期被国外垄断的现状,有望突破国内科研机构和企业在相应技术和应用方面受到的限制,并降低相应的使用成本。
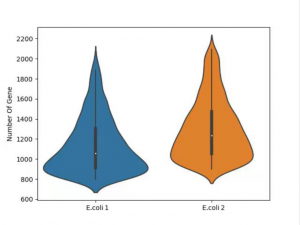
图.单细菌转录组测序测得基因表达数量分布图(纵坐标:单个细菌中检测得到的基因数目;横坐标:样品名称)
敬请期待
M20单细菌测序平台服务
预计将于四月初全面上线!
请关注我们的公众号
以便及时获得相关信息。
如有疑问欢迎致信到我们的邮箱咨询:Info@m20genomics.cn
参考文献
[1]Lan, F., Demaree, B., Ahmed, N., Abate, A.R. (2017). Single-cell genome sequencing at ultra-high-throughput with microfluidic droplet barcoding. Nature biotechnology, 35(7), 640–646.
[2]Blattman, S.B., Jiang, W., Oikonomou, P., Tavazoie, S. (2020). Prokaryotic single-cell RNA sequencing by in situ combinatorial indexing. Nature Microbiology, 5(10):1192-1201.
[3]Kuchina, A., Brettner, L.M., Paleologu, L., Roco, C.M., Rosenberg, A.B., Carignano, A., Kibler, R., Hirano, M., DePaolo, R.W., Seelig, G. (2021). Microbial single-cell RNA sequencing by split-pool barcoding. Science, 371(6531):eaba5257.



