-
Q M20-seq技术原理是?A
M20-seq技术基于随机引物原理,在细胞(核)/细菌内原位逆转录后进行单细胞标记及测序。M20-seq基于随机引物原理可对RNA分子全长进行无偏捕获,此外可以捕获包含lncRNA、CircleRNA在内的非编码RNA。因此,M20-seq技术可在单细胞水平展开更多维度的研究。
-
Q 为什么M20-seq可以进行石蜡包埋样本,RNA降解样本、细菌样本的单细胞检测?A
石蜡包埋样本,RNA降解样本均存在RNA降解情况。经典的mRNA降解通用模型中,Poly(A)尾变短(即脱腺苷化)时, poly(A)结合蛋白PABPC从mRNA上释放下来,mRNA随即被降解。而传统单细胞技术基于Poly(A)进行RNA捕获,因此对RNA完整度要求较高,对石蜡包埋样本,RNA降解样本效果较差。
而细菌RNA本身不具有 poly(A)结构,因此传统单细胞技术无法检测。
M20-seq技术基于随机引物原理,即使RNA分子有一定程度降解、没有poly(A)结构,仍可捕获RNA分子后进行细胞标记,因此可以进行石蜡包埋样本,RNA降解样本、细菌样本的单细胞检测。
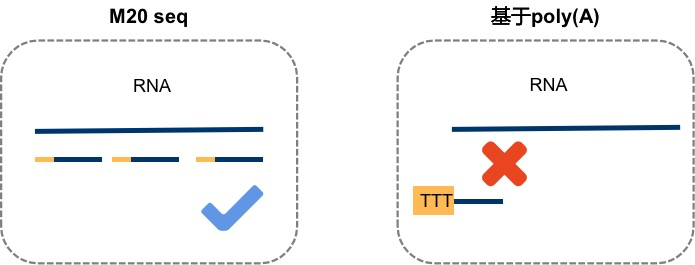
M20-seq与基于poly(A)单细胞技术对比图 -
Q 基于M20-seq技术的VITA单细胞全长转录组平台包含哪几部分?A
VITA单细胞全长转录组平台包含:VITAPilote系列试剂盒、VITACruiser单细胞制备仪和VITASeer软件,均为M20 Genomics自主研发。 -
Q VITA平台实验流程是?A
实验流程如下:
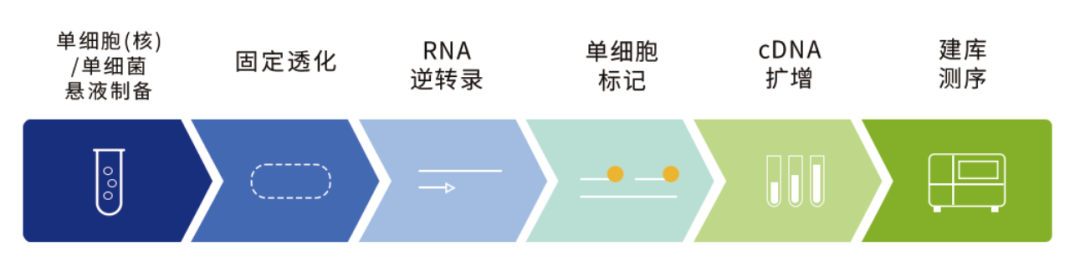
实验流程图
-
Q 整个实验流程耗时如何,哪些步骤可以暂停实验?A
从样本解离至文库构建完成时长约1.5-2.5天。
实验可暂停步骤:细胞核提取后、RNA逆转录后、cDNA延伸后、cDNA扩增后。
-
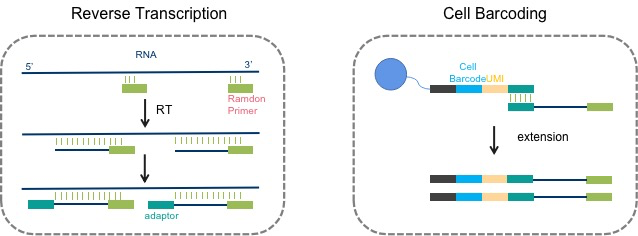
Q 不基于poly(A)捕获,如何进行单细胞标记?A
原位逆转录结束后,在cDNA末端加上捕获接头,捕获接头可与微珠上的oligo序列互补配对。配对后进行延伸使得cDNA链加上细胞标签,完成单细胞标记。

单细胞标记示意图 -
Q 仪器设备的性能如何?A
仪器设备性能详情如下:
- 细胞通量范围:1000-20000个细胞/通道(标准试剂盒);
- 样本通量:4个通道;
- 细胞大小:无限制,细胞、细胞核均可;
- 细胞捕获率:50%~60%;
- 双胞率:每5000个细胞,低于2%;
- 上机时间:不超过10分钟;
- 适用样本类型:新鲜,冻存,石蜡包埋(FFPE);
- 适用物种:真核生物(人、小鼠、大鼠、植物等),原核生物(细菌等);
-
Q VITA平台数据是否兼容其他单细胞分析软件?A
第一步需要使用VITAseer进行数据处理,后续兼容其他单细胞分析软件。

用户登陆

用户注册

用户注册
